本文章介绍了EEG和EMG数据的预处理,由于目的是对EEG和EMG数据在同一时间尺度上的相关性,所以 其中采集的原始数据为TXT格式,且EMG和EEG数据存放在一起。直接使用的图形界面在此:Github Repo: EEG_EMG Process
本文为单一数据的处理,使用我写的图形UI界面则可以批量处理数据。自动从文件里查找数据进行处理。
在进行EEG数据预处理之前,了解数据的基本格式是关键。以下是采集的EEG和EMG数据的格式示例:
1 | # MIX|None|0+True+胫骨前肌+1000|1+True+腓骨长肌+1000|2+True+腓肠肌内侧+1000|3+True+腓肠肌外侧+1000|4+True+股直肌+1000|5+True+股内侧肌+1000|6+True+股二头肌长头+1000|7+True+半腱肌+1000|8+False+胫骨前肌+1000|9+False+腓骨长肌+1000|10+False+肠肌内侧+1000|11+False+肠肌外侧+1000|12+False+EMG13+1000|13+False+EMG14+1000|14+False+EMG15+1000|15+False+EMG16+1000|0+True+P4+80|1+True+CP2+80|2+True+FC5+80|3+True+C3+80|4+True+P3+80|5+True+C2+80|6+True+FC6+80|7+True+C4+80|8+True+CP6+80|9+True+F3+80|10+True+FC2+80|11+True+FC1+80|12+True+F4+80|13+True+CP5+80|14+True+C1+80|15+True+CP1+80 |
MIX|None|:描述了数据类型(例如MIX可能是混合信号)。X+True/False+Channel+SampleRate:表示不同通道的信息,包括是否启用(True/False)、通道名称(如胫骨前肌、P4等)、以及采样率(单位Hz)。#开头的时间点数据,如 13425+24732+39459...。表示实验中的打点信息。但是本次实验中没有用上,因为打点不准确若使用我写的图形界面进行处理,则无需关心,此步骤自动实现。
首先使用MATLAB从文件中读取原始数据,可以采用以下代码:
1 | fid = fopen(filepath); |
根据实验需求,分别提取EEG数据和EMG数据。其中EMG是1-8列,EEG是17-32列
1 | EMGData = [datafile{1:8}]; |
1 | function [EMGData, EEGData] = filterData(EMGData, EEGData) |
由于数据打点信息缺失,或者打点数据不够精确,所以需要手动修正打点数据。所以要先把图画出来,根据图找出精确的位置。
1 | N1 = length(EMGData(1,:)); % EMG通道数量 |
获取到打点信息可以手动补回原来的位置。或者另外存进去。
需要手动构造需要Event 信息文件
事件信息文件使用 纯文本格式(.txt),每一行为一个事件记录,各字段以空格、逗号或制表符分隔。推荐使用**制表符分隔 (tab-separated)**,方便解析。
| 字段名 | 数据类型 | 必需 | 描述 |
|---|---|---|---|
type |
字符串 | 是 | 事件类型标识符,例如 'Stimulus' 或 'Response'。 |
latency |
数字 | 是 | 事件在 EEG 数据中的采样点位置(单位:采样点)。 |
duration |
数字 | 否 | 事件持续时间,以采样点为单位,若未知可填 0。 |
epoch |
数字 | 否 | 所属分段编号,仅当 EEG 数据是分段形式时需要填写。 |
urevent |
数字 | 否 | 链接到源事件数据的编号,用于溯源原始事件。 |
文件可以简单存储为 events.txt,例如:
1 | type latency duration |
在这个步骤直接把数据,依据打点信息,和每个事件的时长,进行剪切,只留下有用的部分。
1 | EEG = pop_importdata('dataformat','ascii','nbchan',0,'data',filepath,'setname',eventFileName,'srate',1000,'pnts',0,'xmin',0,'chanlocs',locs_path); |
pop_importdata
代码参数说明
| 参数名 | 示例值 | 作用说明 |
|---|---|---|
dataformat |
'ascii' |
数据格式类型,'ascii' 表示数据以文本格式存储;其他值可以是 'matlab' 或原生格式。 |
nbchan |
0 |
数据的通道数,0 表示自动检测通道数(常用);您也可以根据实际情况直接填写通道数量。 |
data |
filepath |
数据文件路径,包含实际信号数据的文件路径变量;即每一行是一个时间点,列对应通道数据。 |
setname |
eventFileName |
数据集的名称,显示在 EEGLAB 图形界面中,用于识别导入的 EEG 数据集。 |
srate |
1000 |
数据采样率,单位为 Hz。例如,1000 表示每秒采样 1000 次。 |
pnts |
0 |
每个通道的采样点数,0 表示从文件中自动检测。 |
xmin |
0 |
数据起始时间(以秒为单位),0 表示时间从第一个采样点开始。 |
chanlocs |
locs_path |
通道位置信息文件路径,常用 .loc 或 .ced 文件定义通道的空间位置(头皮电极的位置映射)。 |
其中我的chanlocs文件长这样:
1 | Number labels theta radius X Y Z sph_theta sph_phi sph_radius type |
注意注意: 原来的文件不长这样。但是直接讲原始的导入EEG会有错误,位置回旋转90度,需要每次导入的时候手动修正。或者修正完后导出修正后的位置图,存下来,后续每次加载使用。
使用代码绘制:EEGLAB 提供的函数, EEG 时域波形图 (pop_plotdata)
1 | pop_eegplot(EEG, 1, 1, 0); % 绘制数据的图形 |
EEG: EEGLAB 的数据结构,包含导入的数据。1(连续信号模式): 绘制原始连续信号。1(频道索引标注): 在图上显示通道标注。0(分离窗口): 是否将每个通道分开显示在单独的窗口中。0表示在一个窗口显示所有通道。在图形界面进行绘图
根绝图判断哪个通道的数据损坏,判断方法如下:
数据波形异常
地理不一致性
查看电极分布的头皮地图,损坏的通道通常在头皮拓扑图上呈现孤立的高/低信号区域,与相邻通道不一致。
用代码进行插值:
1 | EEG_interp = pop_interp(EEG, selectedIndices, 'spherical') |
参数解释:
EEG: 这是你原始的 EEG 数据结构,包含了所有信号和信息selectedIndices: 一个包含索引的数组,对应了需要进行插值的电极。'spherical': 这个参数指定了插值方法。在这里,选择了 'spherical' 插值方式。它是一种通过球面坐标系统进行插值的方法,适合当电极的位置遵循球面几何结构时。这种插值方法会考虑到电极的三维空间布局,依据其在球体表面的空间关系来进行插值,而不是简单地用线性方式连接相邻电极的值。手动插值
选择需要插值的通道:
注意:需要一次性对所有的通道进行插值,不可以分多次进行
使用如下代码执行ICA
1 | EEG = pop_runica(EEG, 'icatype', 'runica', 'extended', 1, 'interrupt', 'on', 'chanind', EEG_chans, 'pca', restLen); |
参数解释:
icatype, runica:使用 RunICA 算法进行独立成分分析。extended, 1:启用扩展模式(增加维度,适用于多通道数据)。interrupt, on:允许中断 ICA 计算。chanind, EEG_chans:指定参与 ICA 计算的通道(因为EEG,EMG数据存在一起,所以要选出来EEG)。pca, restLen:使用 PCA 对数据进行降维,restLen 表示主成分数(EEG电极数量-插值过的坏导数量)。该步骤的目标是从 EEG 数据中提取独立成分,以便去除如眼动、肌电等伪影,留下更多反映神经信号的成分。
或者使用手动的方式:
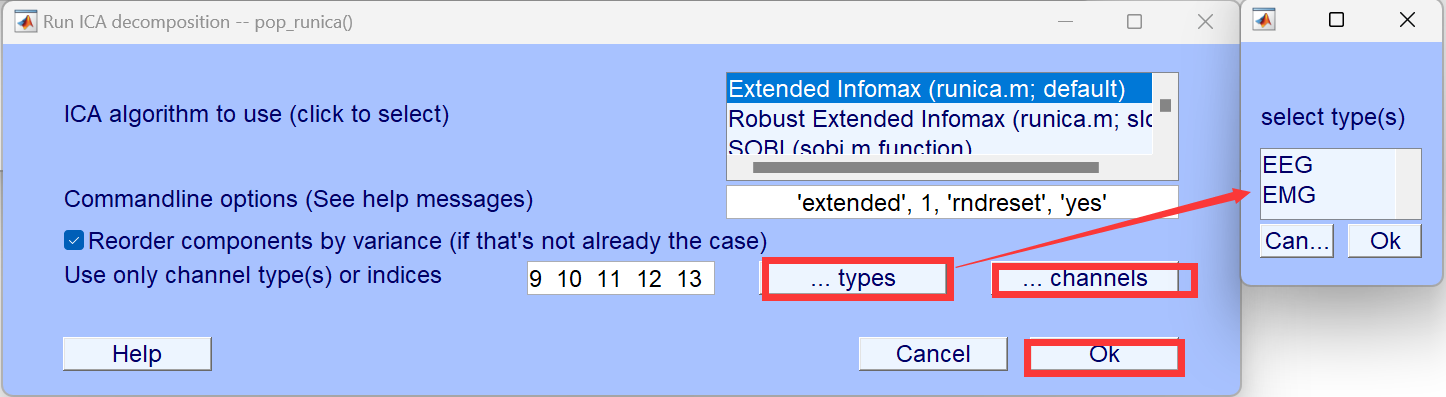
选择EEG数据可以通过图中两种方式,前提是我已经给我的通道进行了类别标记。
执行过程中会弹出窗口,可以随时interrupt中断。
给数据打上标签,对各种伪影类别进行标记
1 | EEG_temp = EEG; % 使用一个临时的 EEG 数据避免直接修改原始数据 |
手动执行: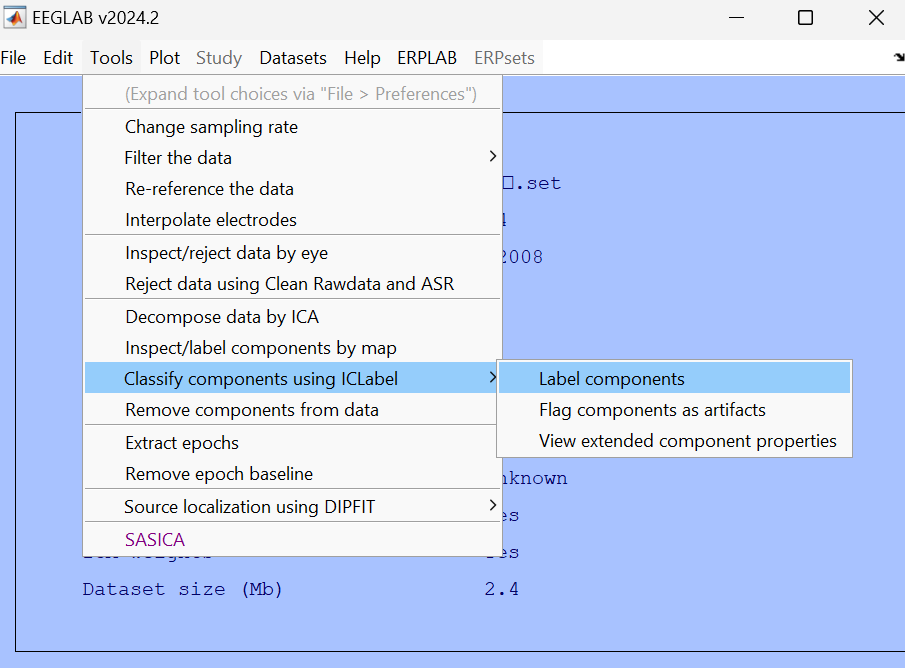
标记完之后会弹出来标记好的内容,这个图没什么特别的用处可以直接关掉,是自动分析伪影之后画出来的分类。
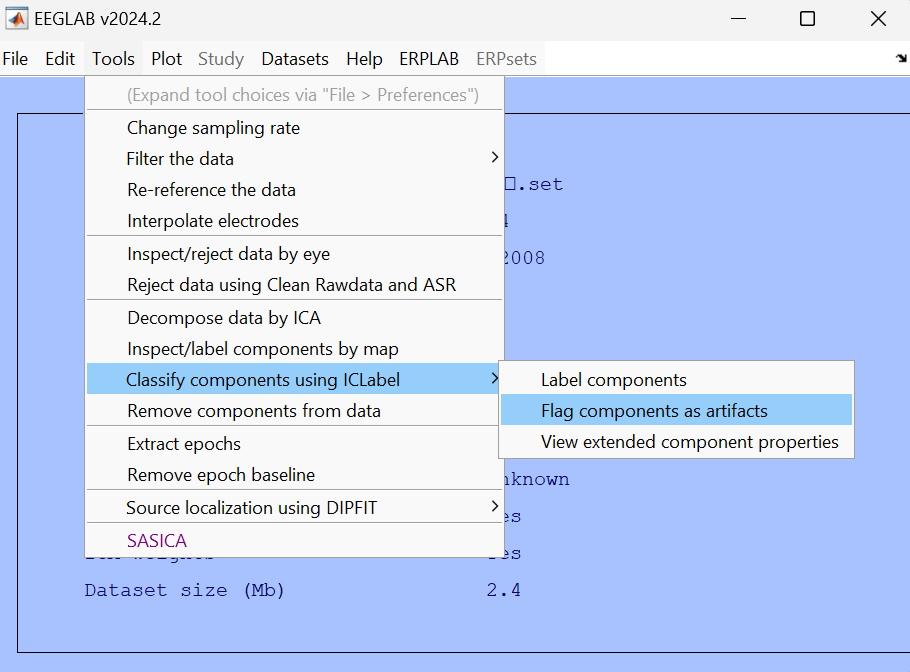
然后使用flag components as artifact功能,自动标记伪影
让给不同的类别设置阈值
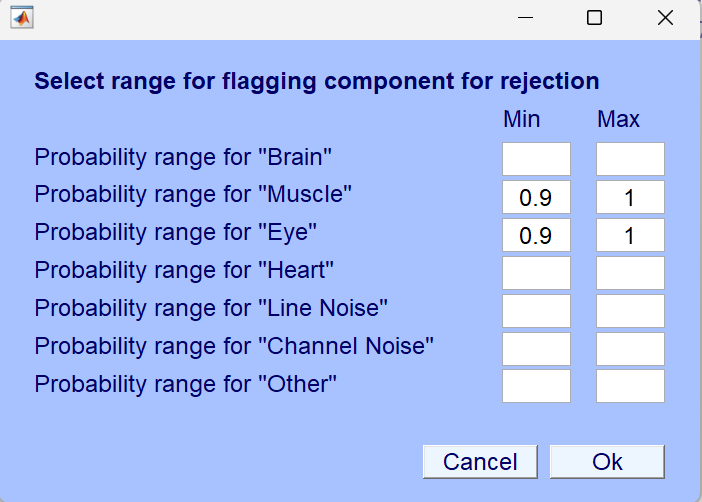
根据自己的数据设置需要的阈值。我自己的数据设置的是每个类别都是0.55,因为我的数据干扰因素很多。
1 | rejected_comps = find(EEG_temp.reject.gcompreject > 0); |
至此,所有的数据处理部分都已经完成了。

